El coronavirus, conocido por los científicos como SARS-CoV-2, ha infectado a más de 3.5 millones de personas en todo el mundo y ha causado más de 250,000 muertes por COVID-19 desde su descubrimiento a fines del año pasado.
NOTA: SARS-CoV-2 es el coronavirus. COVID-19 (Corona Virus Disease 2019) es la enfermedad causada por el virus.
El equipo del Laboratorio Nacional de Los Álamos (Nuevo México), asistido por científicos de la Universidad de Duke y la Universidad de Sheffield en Inglaterra, han identificado 14 mutaciones. Esas mutaciones ocurrieron entre los casi 30,000 pares de bases de ARN que otros científicos dicen que forman el genoma del coronavirus. Los autores se han centrado en una mutación llamada D614G, que es responsable del cambio en la proteína S (spike o espiga) del COVID-19 virus. La mutación no solo hace que el virus sea más contagioso, sino que también puede causar que las personas que ya han tenido COVID-19 sufran un segundo ataque de la enfermedad.
El estudio se basó en un análisis computacional de más de 6,000 secuencias de coronavirus de todo el mundo, recopilado por la Iniciativa Global para Compartir Todos los Datos de Influenza (GISAID), una organización público-privada en Alemania. Una y otra vez, el análisis encontró que la nueva versión estaba en transición para convertirse en dominante.
La mutación Spike D614G es motivo de preocupación urgente; comenzó a extenderse en Europa a principios de febrero, y cuando se introdujo en nuevas regiones, rápidamente se convirtió en la forma dominante. Además, presentan evidencia de recombinación entre cepas que circulan localmente, indicativas de infecciones por cepas múltiples. Estos hallazgos tienen implicaciones importantes para la transmisión del SARS-CoV-2, la patogénesis y las intervenciones inmunes.
Hasta la fecha 13 Abril 2020, han identificado catorce mutaciones en la proteína Spike que se que se acumulan a medida que el virus se propaga. Pero una mutación en particular preocupa a los investigadores, la mutación Spike D614G, que está reemplazando la forma original del virus Wuhan de forma rápida y repetida en todo el mundo.
Los investigadores dicen que el nuevo apareció en febrero en Europa, luego en los Estados Unidos en la costa este, y ha sido la cepa dominante en todo el mundo desde mediados de marzo. Infectando a muchas más personas que las cepas anteriores que salieron de Wuhan, China. En algunas naciones, era la única cepa prevalente. Los investigadores no saben por qué la nueva cepa es más infecciosa que sus predecesoras.
Para un virus, propagarse durante unos meses y alcanzar más de 3 millones de huéspedes, es un largo período de tiempo para evolucionar y cambiar.
Hasta ahora, se estima que el SARS-CoV-2 tiene una tasa de menos de 25 mutaciones por año, en comparación con las 50 de la influenza.
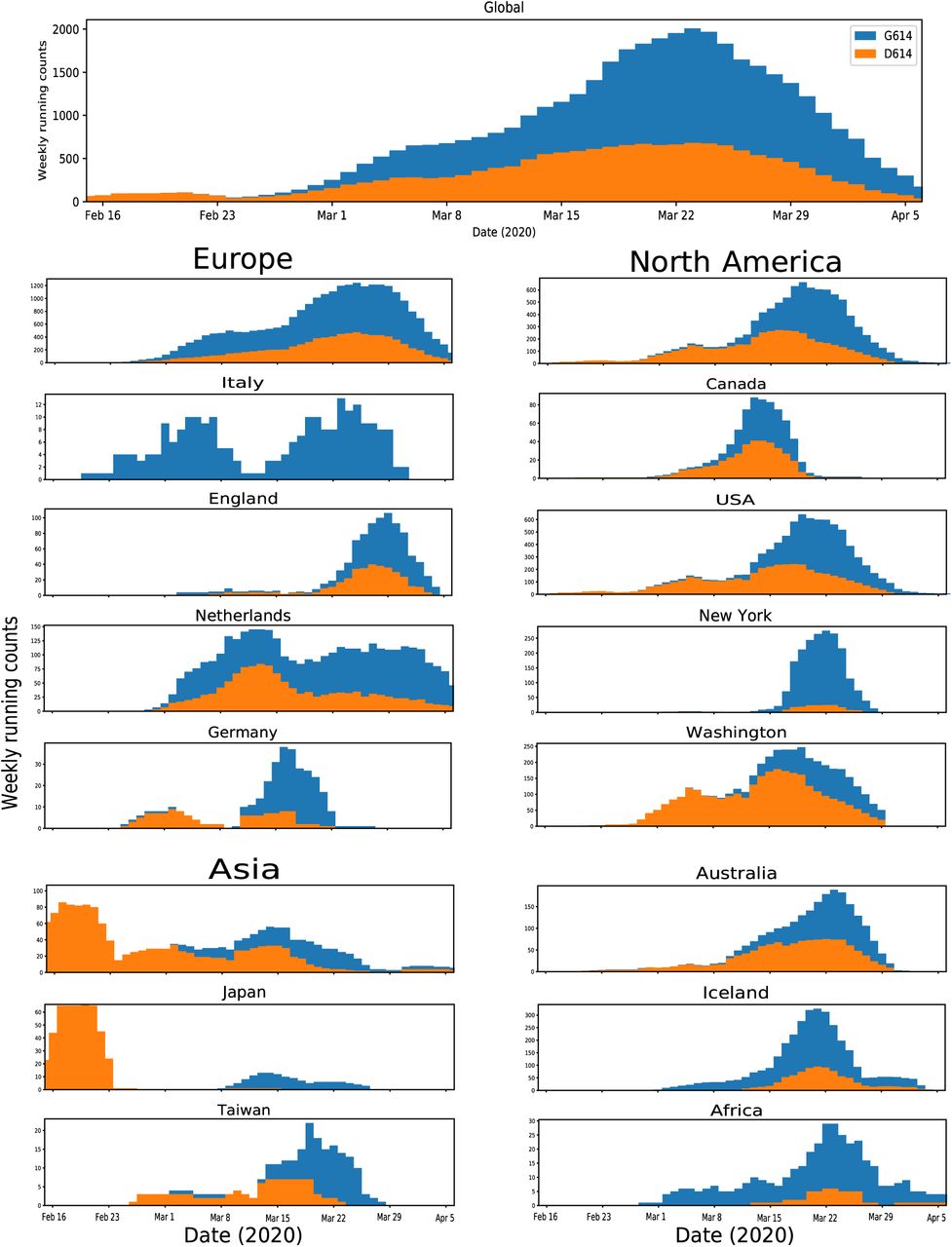
En casi todos los casos, poco después de que G614 (azul) ingresa a una región, comienza a dominar la muestra. Forma original de Wuhan (D614, naranja),
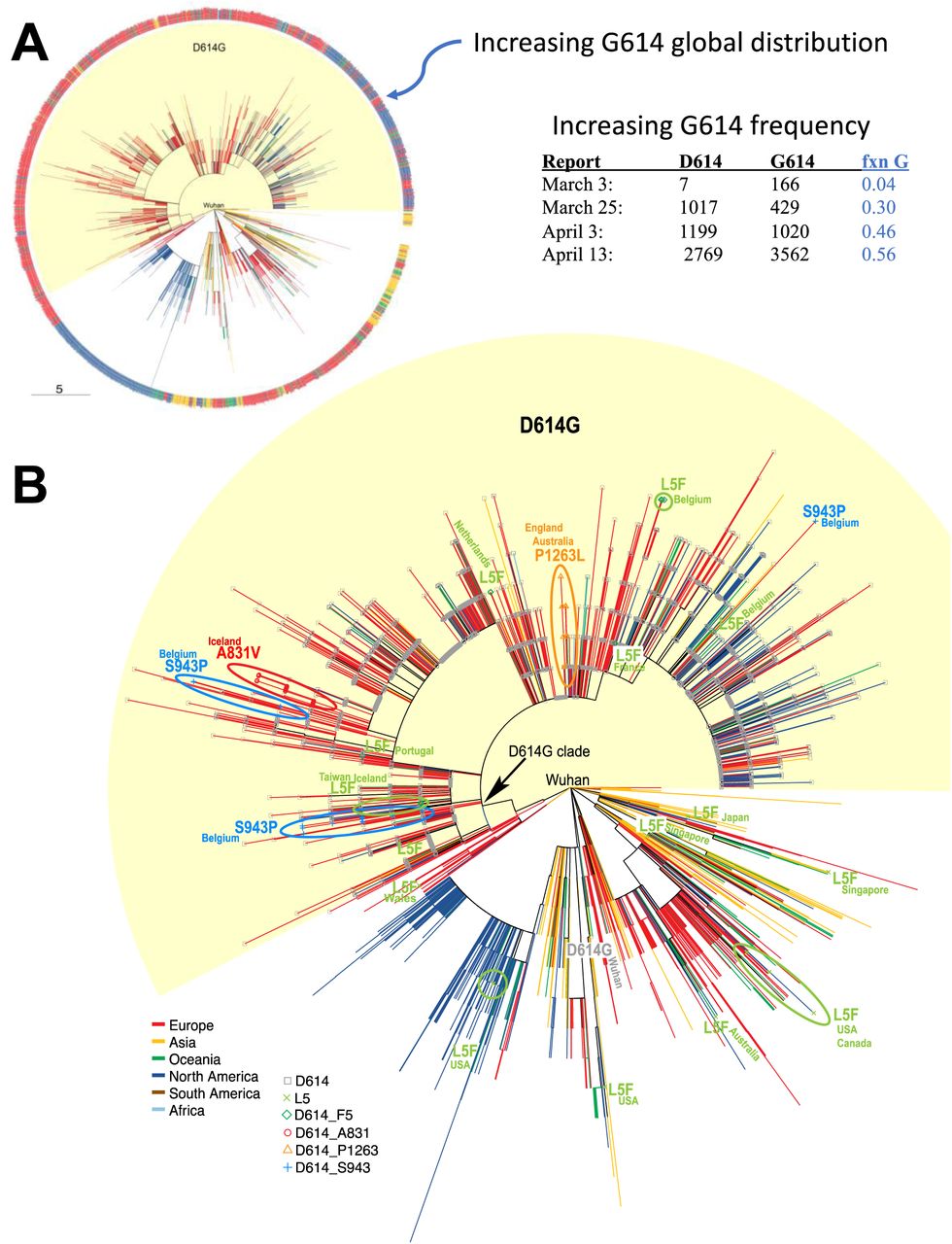
Árboles filogenéticos basados en 4.535 alineamientos recortados del SARS-CoV-2del genoma completo de GISAID.
Las regiones del mundo donde se muestrearon las secuencias se indican mediante colores. A mediados de abril, G614 era más común que la forma D614 original aislada de Wuhan, y en lugar de estar restringida a Europa (rojo), había comenzado a extenderse a nivel mundial. El mismo árbol se expandió para mostrar patrones interesantes de mutaciones de Spike que estamos rastreando en el contexto del árbol filogenético basado en el genoma completo. Encuentran tres patrones distintos:
- mutaciones que parecen ser predominantemente parte de un solo linaje (P1263L, naranja en el Reino Unido y Australia, y también A831V, rojo, en Islandia);
- una mutación que se encuentra en regiones muy diferentes, tanto geográficamente como en la filogenia, lo que indica que la misma mutación parece surgir y muestrearse independientemente (verde L5F, raro pero encontrado en lugares dispersos en todo el mundo);
- y una mutación en secuencias de la misma ubicación geográfica, pero que surge en linajes muy distintos en la filogenia (S943P), azul, que se encuentra solo en Bélgica
Un huésped sano pero infectado por el coronavirus es un caballo de Troya que va al trabajo, a la escuela, a las reuniones, viaja e interactúa con las personas, mientras que sin querer propaga el virus.
Las mutaciones también pueden hacer que un virus sea más o menos virulento. Una idea común es que la virulencia solo cambiará, ya sea aumentando o disminuyendo, si aumenta la tasa de transmisión del virus, lo que efectivamente significa un aumento en el número de 'descendencia' del virus.
Al referirse a la mutación Spike D614G, William Hanage, un epidemiólogo de Harvard que se especializa en la evolución de enfermedades infecciosas, dijo que 'si hubiera algo que influyera en la transmisibilidad, entonces la proteína Spike (espiga) es el lugar donde esperaría encontrarla'.
CONCLUSIONES
- Estudio preliminar ha identificado 14 mutaciones, pero la mutación llamada D614G es en la que han centrado su investigación.
- Esta mutación D614G, es responsable del cambio en la proteína S (spike o espiga) del COVID-19 virus.
- Y esta mutación no solo hace que el virus sea más contagioso, sino que también pueda causar que las personas que ya han tenido COVID-19 puedan sufrir un segundo ataque de la enfermedad.
- Apareció en febrero en Europa, luego en los Estados Unidos en la costa este, y ha sido la cepa dominante en todo el mundo desde mediados de marzo.
REFERENCIA
Korber B, Fischer WM, Gnanakaran S, et al. Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. bioRxiv preprint this version posted April 30, 2020 . doi: https://doi.org/10.1101/2020.04.29.069054
APÉNDICE
GENOMA, ESTRUCTURA, Y PROTEINA S (Spike o espiga)
El genoma COVID-19 consta de ARN monocatenario contiene 29.891 nucleótidos, que codifican 9.860 aminoácidos
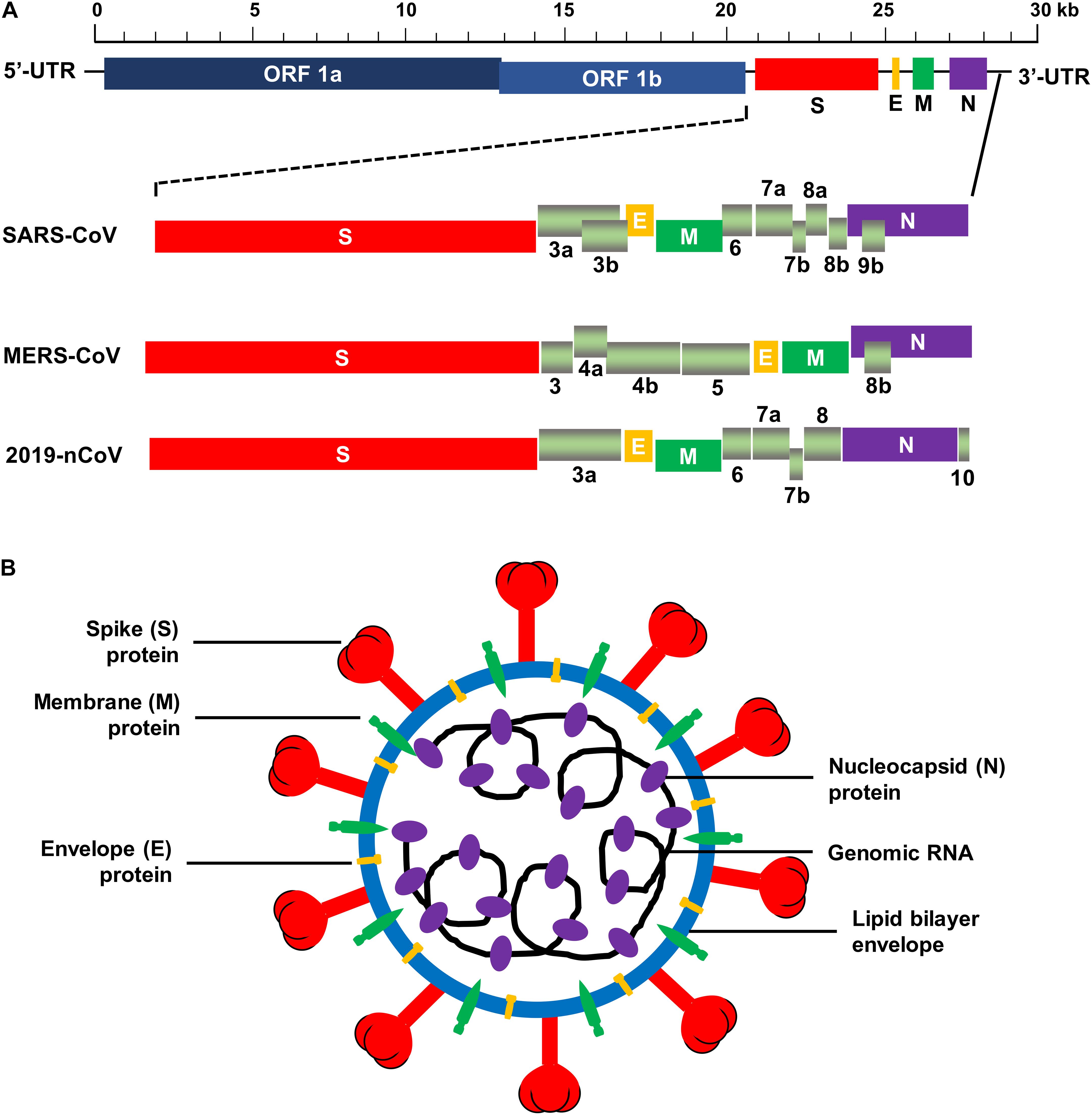
Estructura esquemática de SARS-CoV, MERS-CoV y 2019-nCoV, SARS-CoV-2 o COVID-19.
Se comparan las regiones genómicas o los marcos de lectura abierta (ORF). Proteínas estructurales, incluidas las proteínas de espiga (S), envoltura (E), membrana (M) y nucleocápside (N), así como proteínas no estructurales traducidas de ORF 1a y ORF 1b y proteínas accesorias ), y 3a, 6, 7a, 7b, 8 y 10 (para 2019 -nCoV). 5'-UTR y 3'-UTR, regiones no traducidas en las regiones N y C-terminales, respectivamente. Kb, par kilobase.
La proteína S (Spike o espiga) está muy glicosilada, utiliza una secuencia de señal N-terminal para obtener acceso al retículo endoplásmico (ER) y mediar la unión a los receptores del huésped. Es la estructura más grande y produce los distintos picos en la superficie del virus. Para la mayoría de los coronavirus, la proteína S es escindida por una proteasa similar a la furina de la célula huésped en dos polipéptidos separados S1 y S2.
- Los investigadores han determinado que la parte más variable del genoma del coronavirus se encuentra precisamente en el dominio de unión al receptor de la proteína S, una proteína necesaria para la invasión del virus. En humanos, este dominio proteico tiene una afinidad especial por los receptores ACE2 de las células del huesped
- La proteína S media la entrada viral en las células huésped uniéndose primero a un receptor huésped a través del dominio de unión al receptor (RBD) en la subunidad S1 y luego fusionando las membranas viral y huésped a través de la subunidad S2
- La proteína espiga (proteína S) realiza dos tareas principales que ayudan en la infección del huésped: 1) media la unión entre el virus y los receptores de la superficie de la célula huésped, y 2) facilita la entrada viral en la célula huésped al ayudar en la fusión del virus y membranas de células huésped
PROF. DR. FERNANDO GALAN
