La nueva herramienta CRISPR llamada (PRIME EDITING) edición principal, descrita en un estudio publicado el 21 de octubre de 2019, en Nature,
- Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, Chen PJ, Wilson C, Newby GA, Raguram A, Liu DR. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 2019;576:149-157. doi: 10.1038/s41586-019-1711-4.
David Liu, biólogo químico del Broad Institute of MIT y Harvard en Cambridge, Massachusetts y autor principal del estudio, estima que la edición principal podría ayudar a los investigadores a abordar aproximadamente el 89% de las mutaciones patogénicas de las más de 75,000 variantes de ADN asociadas a enfermedades, que figuran en ClinVar, una base de datos pública desarrollada por los Institutos Nacionales de Salud de EE. UU. La especificidad de los cambios de los que es capaz esta última herramienta también podría facilitar que los investigadores desarrollen modelos de enfermedades en el laboratorio y estudien la función de genes específicos.
Aquí transcribo traducidas, algunas preguntas y respuestas extraídas de un artículo de Sharon Begley, Escritora Senior, Ciencia y Descubrimiento. STAT invitó a los lectores a enviar preguntas sobre la nueva tecnología
- STAT es una compañía de medios enfocada en encontrar y contar historias convincentes sobre salud, medicina y descubrimientos científicos
BREVE INTRODUCCIÓN
Evolución de la edición del genoma
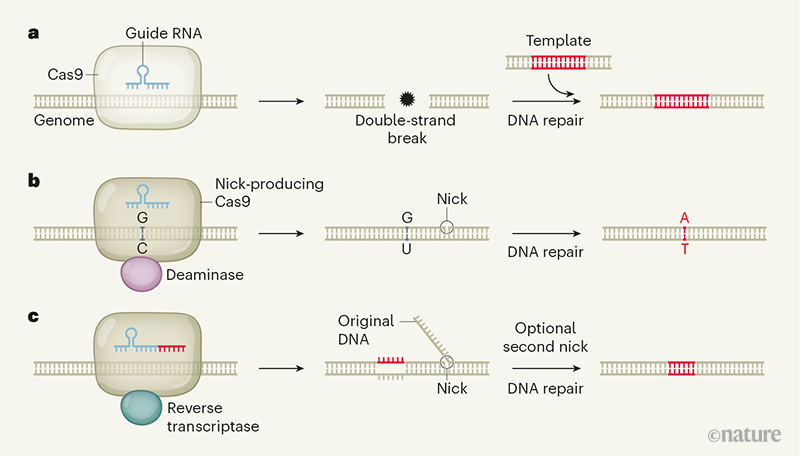
a, En la edición convencional del genoma 2013, una enzima Cas9 es dirigida a una posición en el genoma por un ARN guía, y produce una ruptura de doble cadena. La maquinaria de reparación del ADN de la célula huésped repara la ruptura, guiada por una plantilla de ADN, incorporando secuencias de plantillas en el dúplex.
b, En un enfoque llamado edición de base 2016, una Cas9 que produce solo roturas de cadena única (mellas) funciona con una enzima desaminasa. La desaminasa modifica químicamente una base de ADN específica; aquí, una base de citidina (C) se convierte en uracilo (U). La reparación del ADN luego repara el corte y convierte un intermedio de guanina-uracilo (G – U) en un par de bases adenina-timina (A-T). Este método es más preciso que a, pero solo realiza ediciones de un solo nucleótido.
c, En la edición principal 2019, que puede editar con precisión las secuencias de ADN. Una Cas9 productora de un corte en solo una cadena de ADN, y una enzima transcriptasa inversa producen DNA mellado en el que se han incorporado las secuencias correspondientes al RNA guía. La secuencia de ADN original se corta y la reparación del ADN luego arregla la hebra mellada para producir un dúplex completamente editado. En algunos casos, se realiza otro corte en la hebra no editada del dúplex antes del paso de reparación del ADN (no se muestra).
PREGUNTAS Y RESPUESTAS
¿Podría por favor, comparar los pros y los contras de la edición principal versus la edición de base?
En este sentido, prime editing (Edición principal) supone una mejora respecto a los editores de base que permitían modificar el ADN sin cortar el ADN, pero cuyas modificaciones posibles estaban limitadas a únicamente algunos cambios concretos.
La primera diferencia es que la edición base se ha utilizado ampliamente durante los últimos 3 años y medio en organismos que van desde bacterias a plantas, a ratones y primates.
Si bien estamos muy entusiasmados con la edición principal, es completamente nueva y hasta ahora solo se ha publicado un artículo. Por lo tanto, hay mucho por hacer antes de que podamos saber si la edición principal demostrará ser tan general y robusta como lo ha demostrado ser la edición de bases.
Comparamos directamente editores principales y editores básicos en nuestro estudio, y descubrimos que los editores base actuales pueden ofrecer una mayor eficiencia de edición y menos subproductos independientes que los editores principales, mientras que los editores principales ofrecen más flexibilidad de orientación y mayor precisión de edición.
Entonces, cuando la edición deseada es una mutación de punto de transición (C a T, T a C, A a G o G a A), y la base objetivo está bien posicionada para la edición de base (es decir, existe una secuencia PAM aproximadamente 15 bases del sitio de destino), luego la edición de bases puede resultar en una mayor eficiencia de edición y menos subproductos. Cuando la base de destino no está bien posicionada para la edición de base, o cuando hay otras bases C o A 'espectadoras' cercanas que no deben editarse, la edición principal ofrece grandes ventajas ya que no requiere una secuencia PAM posicionada con precisión y es un verdadera capacidad de edición de 'buscar y reemplazar', sin posibilidad de edición de espectadores no deseados en bases vecinas.
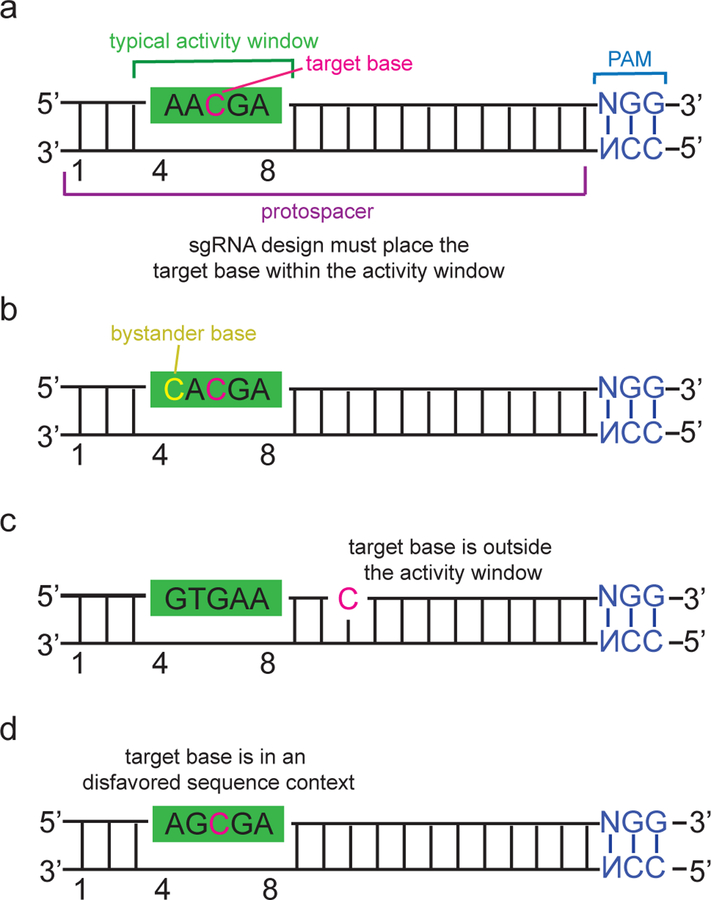
- Un lugar ideal de edición de base de destino. La base objetivo se encuentra dentro de la ventana de actividad del editor de base relativa al sitio PAM, solo hay una base objetivo en la ventana de actividad, y la base objetivo se encuentra en un motivo (AC en este ejemplo) que se desamina eficientemente por la mayoría de las citosinas de editores base.
- Ejemplo de un sitio objetivo con una base como espectadora. Si la edición del espectador (desaminación de la citosina que se muestra en amarillo) no es deseada, se puede usar una ventana estrecha o un editor de base específico del contexto para editar preferentemente la base del objetivo sobre la base del espectador
- La base del objetivo se encuentra fuera de la ventana de actividad. La edición de base en este objetivo puede ser posible con editores de base que reconocen diferentes PAM.
- La base objetivo se encuentra dentro de un contexto de secuencia que puede no ser un sustrato eficiente para una desaminasa particular. La edición del objetivo puede mejorarse utilizando un editor con una desaminasa diferente, o un editor más tolerante al ADN metilado
Por supuesto, para clases de mutaciones que no sean los cuatro tipos de mutaciones puntuales que los editores de base pueden hacer, como inserciones, eliminaciones y los otros ocho tipos de mutaciones puntuales, según nuestro conocimiento, la edición principal es actualmente el único enfoque que puede hacer que estos mutaciones en células humanas sin requerir cortes de ADN bicatenarios o plantillas de ADN separadas
¿Cuál considera es el paso clave en el desarrollo del sistema de edición principal?
Hubo algunos desafíos clave que superar, pero destacaré dos.
La idea general detrás de la edición principal es que encuentra un sitio diana (objetivo) en el genoma y escribe directamente nuevas cadenas de letras de ADN que pueden reemplazar la secuencia de ADN original.
Uno de los primeros y mayores desafíos fue diseñar una forma de hacer una nueva cadena de ADN de nuestra elección en prácticamente cualquier sitio de un vasto genoma. En última instancia, el desarrollo del sistema de edición principal fue posible gracias al conocimiento colectivo de la comunidad científica sobre la bioquímica tanto de CRISPR-Cas9 como de las transcriptasas inversas.
Un segundo gran desafío fue lograr la edición principal en células de mamíferos. El desarrollo inicial se realizó en un tubo de ensayo, con un conjunto intermedio de experimentos realizados tanto en tubos de ensayo como en células de levadura. Si bien los resultados de estos primeros estudios fueron alentadores, el funcionamiento interno de una célula de mamífero es mucho más complejo y menos predecible.
- Nuestros experimentos iniciales con células de mamíferos no produjeron ninguna edición, y no estaba claro si el problema era uno que pudiéramos superar o si era una característica intrínseca de las células de mamíferos.
El fortalecimiento del complejo cebador-plantilla resultó en la primera edición primaria de bajo nivel pero inequívoca en células humanas, validando que una célula de mamífero podría soportar todos los pasos clave.
¿Hay algún tipo de células en la que sea más difícil usar la edición principal?
Los cinco tipos de células en las que hemos realizado una edición principal hasta el momento (cuatro líneas celulares de cáncer humano y neuronas corticales de ratón primarias posmitóticas) son un pequeño subconjunto de los cientos de tipos de células que los investigadores usan en las ciencias biomédicas. Nuestra comprensión de los factores celulares que determinan la eficiencia de la edición principal es incompleta en este momento, por lo que es difícil predecir qué células serán mejores o menos adecuadas para la edición principal. Dicho esto, la capacidad de introducir la construcción del editor principal (en forma de ADN, ARN y / o proteína) en las células es ciertamente un factor que contribuye a la facilidad o dificultad de usar cualquier agente de edición del genoma en un tipo de célula.
¿Cuáles son los alelos patogénicos del 11% que están teóricamente fuera del alcance de esta tecnología?
Para calcular la fracción de las más de 75,000 mutaciones patogénicas en ClinVar que, en principio, pueden corregirse con la edición principal, sumamos cada una de las clases de cambios que realizamos en nuestro estudio:
- los 12 posibles cambios de base a base,
- las inserciones tan grandes como 44 pb,
- deleciones tan grandes de 80 pb,
- y combinaciones de las anteriores,
produciendo aproximadamente el 89% de las mutaciones patogénicas en ClinVar.
- Es importante tener en cuenta que, al contrario de algunas citas erróneas de nuestro artículo por parte de algunos analistas de Twitter y medios de comunicación, esto no es lo mismo que decir que el 89% de las enfermedades genéticas pueden tratarse: el tratamiento de una enfermedad genética generalmente requiere muchos componentes, solo uno de ellos es corregir la mutación que causa la enfermedad.
El 11% restante de alelos patógenos en ClinVar que no contamos en el total del 89% son los clasificados como:
- 'ganancia de número de copia',
- 'pérdida de número de copia' y 'otro'.
Aunque sospecho que la edición principal podría mejorar. Algunos de los efectos de estas mutaciones, dado que no sabemos qué tan grande puede agregar o eliminar un segmento de la edición principal de ADN, para ser conservadores, no incluimos ninguna mutación dentro de estas categorías como potencialmente corregible con la edición principal.
¿Cómo se realizan ediciones en un número suficiente de células para lograr una cura?
Esta es una pregunta importante, que tiene respuestas muy diferentes según la enfermedad. Para algunas enfermedades, como ciertas afecciones de la sangre, las células relevantes pueden editarse fuera del cuerpo (ex vivo), luego trasplantarse nuevamente al paciente, lo que facilita la edición de las células relevantes en algunos aspectos.
Las enfermedades genéticas también difieren en la fracción de células diana que deben corregirse para mejorar o incluso curar la enfermedad. Afortunadamente, para muchas (pero no todas) enfermedades genéticas, se cree que la corrección de incluso una fracción modesta de células diana ofrece beneficios a los pacientes. Determinar la fracción de células editadas que dará como resultado un beneficio clínico es uno de los requisitos
¿Se puede utilizar una edición principal en genomas mitocondriales?
Edición de ADN mitocondrial con CRISPR es una vieja aspiración en este campo, pero plantea problemas especiales asociados, a como introducir la guía de ARN en la mitocondria.
La entrega de máquinas moleculares grandes, como los agentes de edición del genoma a las células objetivo es un problema ¿Cuándo se pondrá al día el campo de los métodos de entrega virales y no virales de esta entrega, especialmente para los no virales?
Es cierto que las máquinas moleculares más sofisticadas con componentes más o más grandes requieren la entrega de cargas útiles más grandes, pero algunos hechos son útiles para mantener una perspectiva adecuada. Las instrucciones que codifican la forma más comúnmente utilizada de CRISPR-Cas9 más una guía de ARN y las secuencias reguladoras asociadas necesarias para producirlas en células humanas suman alrededor de 5,000 pb (pares de bases) de secuencia de ADN.
- Los editores base más un ARN guía y secuencias reguladoras asociadas son alrededor de 6,000 pares de bases.
- Y los editores principales más un ARN guía y secuencias reguladoras asociadas son alrededor de 7,000 pares de bases.
Entonces, si bien los editores principales son más grandes que los editores base, que a su vez son más grandes que el CRISPR Cas9 original, sus diferencias de tamaño no son tan dramáticas como lo han implicado ciertos informes de los medios. Ya se han descrito varias variantes de Cas9, variantes de desaminasa y transcriptasa inversa que podrían reducir sustancialmente el tamaño de las nucleasas programables, los editores de base y los editores principales , y nosotros y otros ya hemos comenzado a usar estas variantes para minimizar el tamaño de las instrucciones del agente de edición del genoma.
Mi pregunta está relacionada con esta información en el artículo, '... El tercer componente, una enzima llamada transcriptasa inversa que se fusiona con Cas9, copia los nucleótidos de ARN transportados por el pegRNA y los transforma en nucleótidos de ADN, que reemplazan a los del sitio objetivo. ¿Cómo hace esto la transcriptasa inversa? ¿De dónde obtiene los nucleótidos que se convierten en la parte fija / dirigida del ADN? ¿Están los nucleótidos necesarios flotando en el núcleo o en el citosol y, de ser así, cómo se conectan con el pegRNA?
La enzima transcriptasa inversa en los editores principales usa los mismos bloques de construcción de ADN, llamados trifosfatos de desoxinucleótidos (dNTP), que la célula utiliza naturalmente para su propia replicación y reparación de ADN.
Por lo tanto, estos bloques de construcción son abundantes en la célula y están disponibles para su incorporación en nuevas cadenas de ADN por la transcriptasa inversa. La enzima transcriptasa inversa usa la secuencia de pegRNA, una cadena de nucleótidos de ARN, para moldear la nueva cadena de nucleótidos de ADN, y agrega la nueva secuencia de ADN, letra por letra, a la cadena de ADN objetivo. Durante el proceso, la transcriptasa inversa verifica el emparejamiento correcto de nucleótidos para garantizar que coincida con la plantilla de pegRNA
TRANSCRIPTASA INVERSA
ARN → TRANSCRIPTASA INVERSA → ADN CONPLEMENTARIO (ADNc)
El Sistema Nervioso Central (SNC) parece el área con menos soluciones. ¿Cuándo espera ver la edición de genes para enfermedades monogénicas del SNC (Rett, Angelman, Síndrome de Prader-Willi, etc.)? ¿Y para las enfermedades poligénicas del SNC (autismo, Parkinson, Alzheimer, demencia, etc.)?
Existe un gran interés en la aplicación de métodos de edición de genes, como la edición principal, para el tratamiento de enfermedades que actualmente tienen pocas o ninguna opción terapéutica efectiva.
- Desafortunadamente, las enfermedades del sistema nervioso central son ejemplos comunes de esta categoría.
- Se deben abordar una serie de desafíos y preguntas antes de que un sistema terapéutico de edición de genes basado en la edición principal pueda usarse en pacientes con trastornos del SNC, incluida la entrega del agente de edición a las células correctas, y determinar el alcance de la edición y el plazo para edición que se requiere para lograr un efecto terapéutico.
Es importante enfatizar que, por lo general, lleva años realizar los experimentos necesarios en células y modelos animales de una enfermedad genética determinada antes de que una terapia humana potencial que use la edición principal, o cualquier otra tecnología de edición del genoma, pueda considerarse lista para ensayos clínicos.
RESUMEN
- Es importante tener en cuenta que, al contrario de algunas citas erróneas de nuestro artículo por parte de algunos analistas en Twitter y medios de comunicación, esto no es lo mismo que decir que el 89% de las enfermedades genéticas pueden tratarse: el tratamiento de una enfermedad genética generalmente requiere muchos componentes, solo uno de ellos es corregir la mutación que causa la enfermedad
- Edición de ADN mitocondrial con CRISPR es una vieja aspiración en este campo, pero plantea problemas especiales asociados, a como introducir la guía de ARN en la mitocondria.
- Es importante enfatizar que, por lo general, lleva años realizar los experimentos necesarios en células y modelos animales de una enfermedad genética determinada antes de que una terapia humana potencial que use la edición principal, o cualquier otra tecnología de edición del genoma, pueda considerarse lista para ensayos clínicos.
PROF. DR. FERNANDO GALÁN
