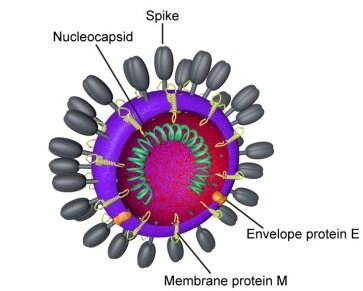
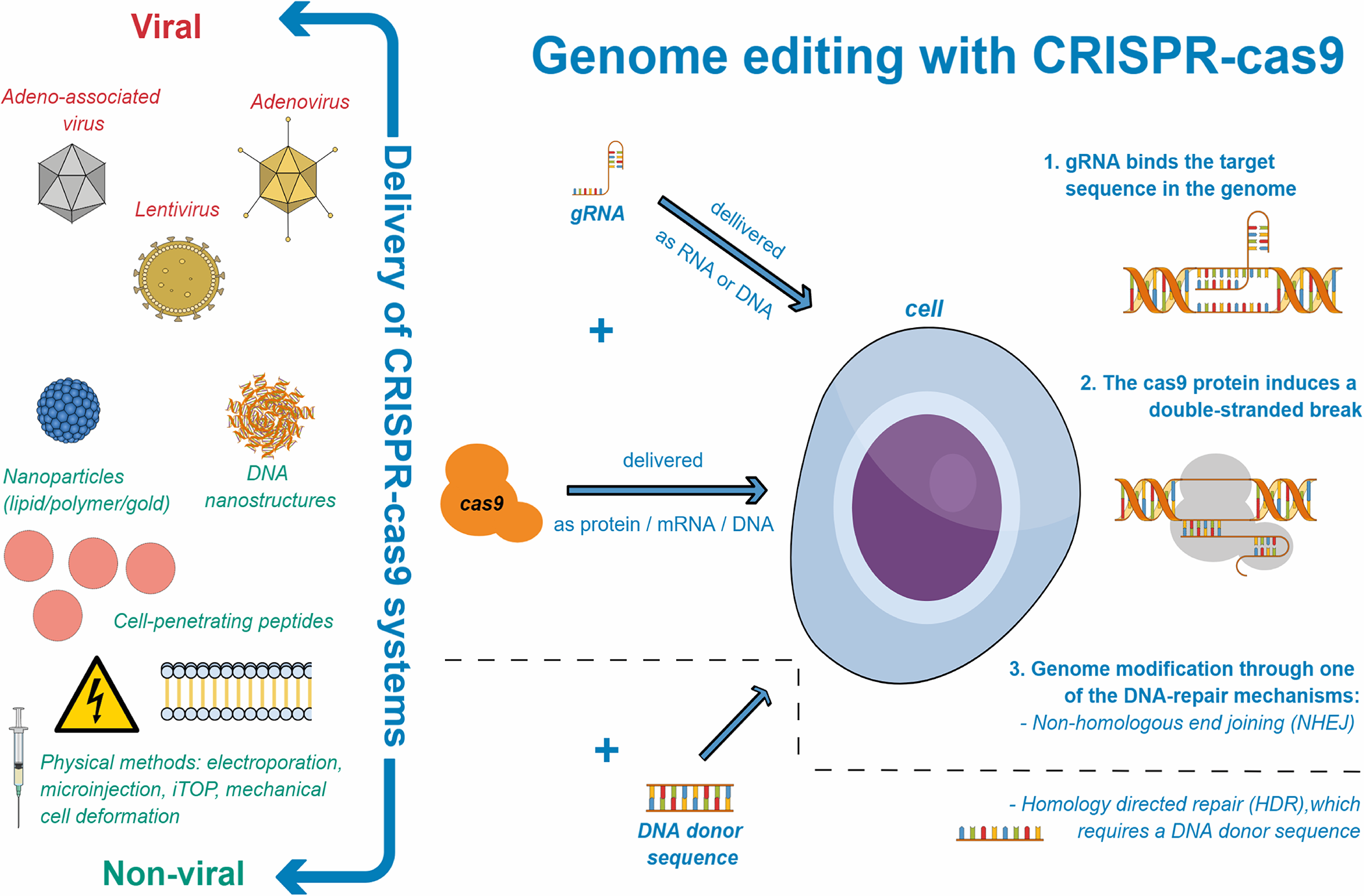
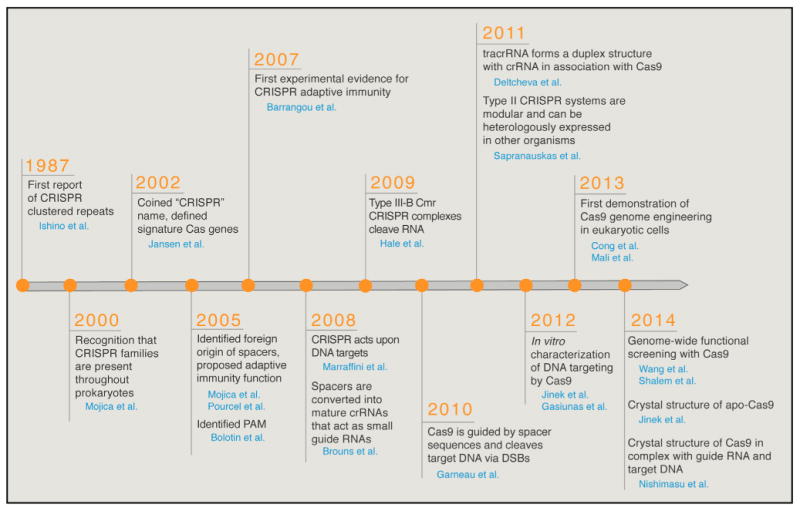
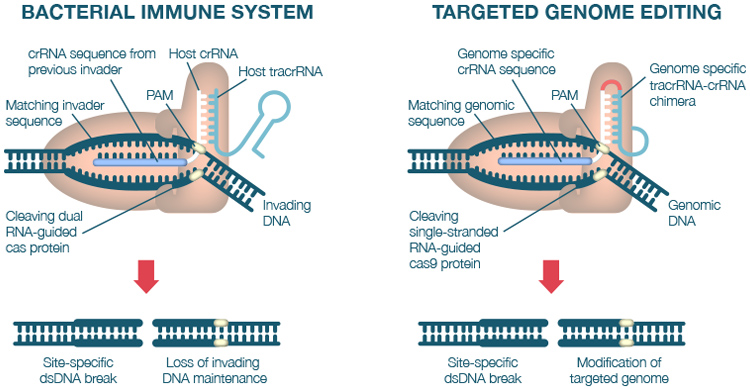
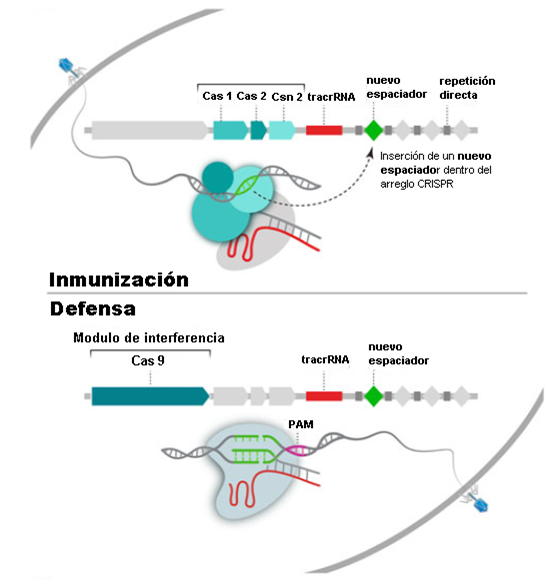
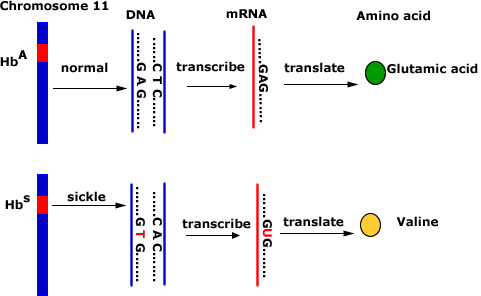
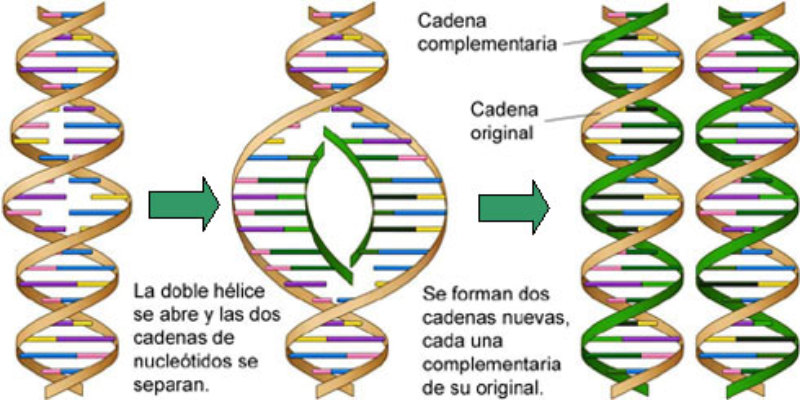
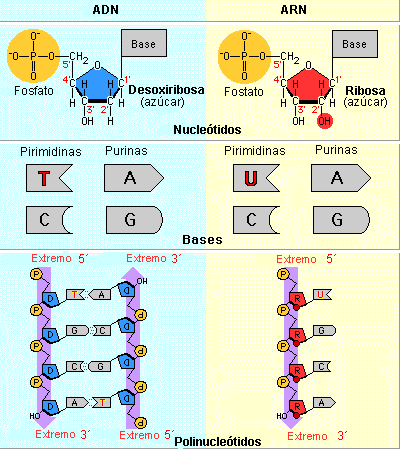
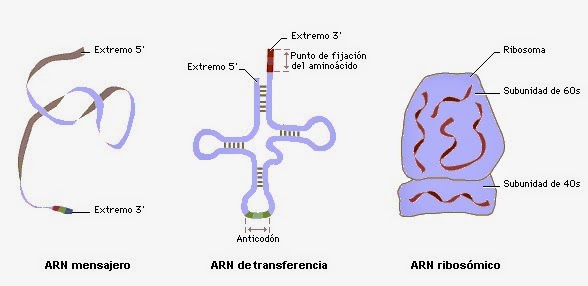
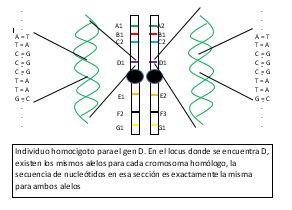
Los rumores, los mitos y la información errónea sobre el nuevo coronavirus se han extendido tan rápido como el virus mismo.
La gripe común causa hasta 5 millones de casos de enfermedades graves en todo el mundo y mueren 650,000 personas cada año, según la Organización Mundial de la Salud.
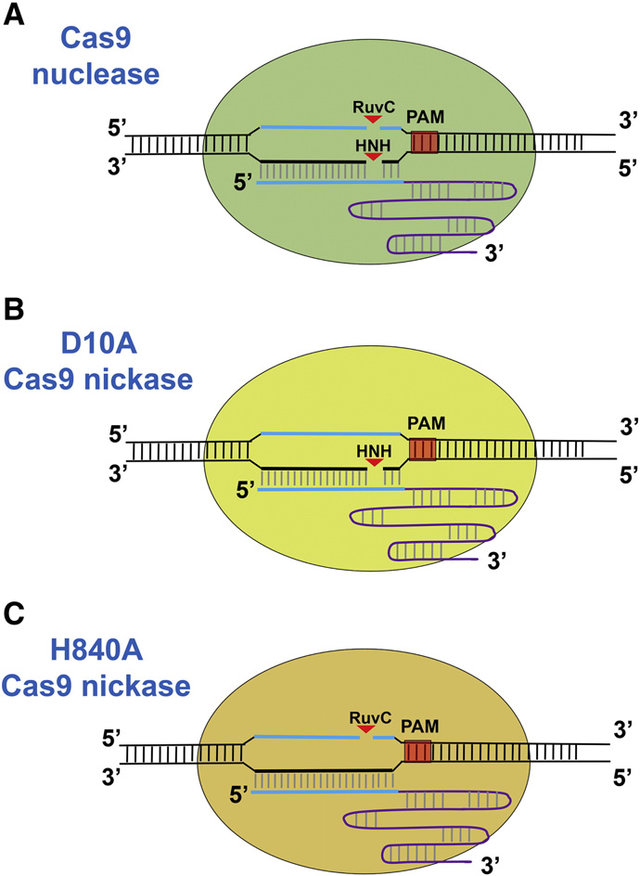
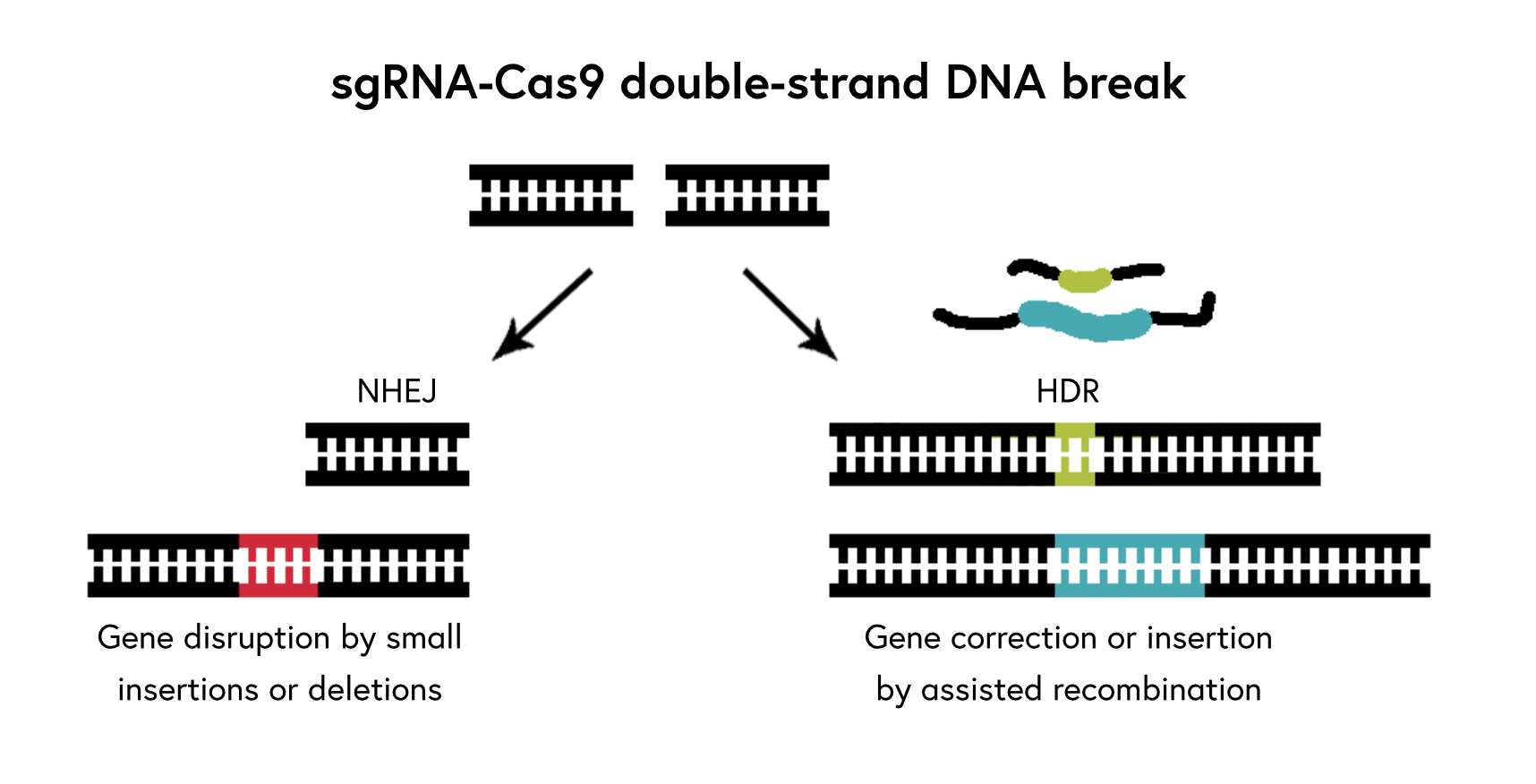
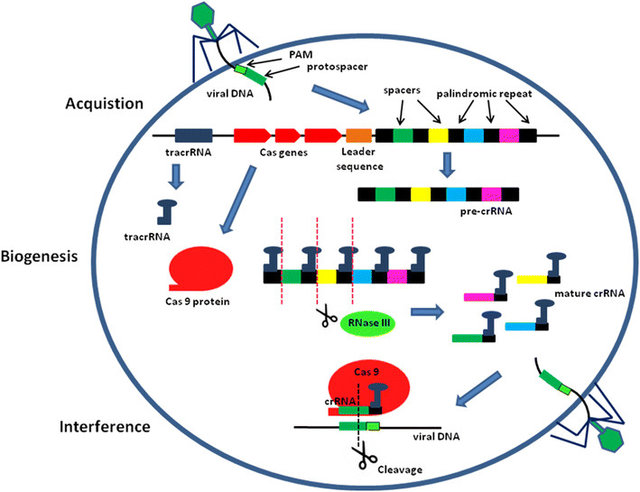
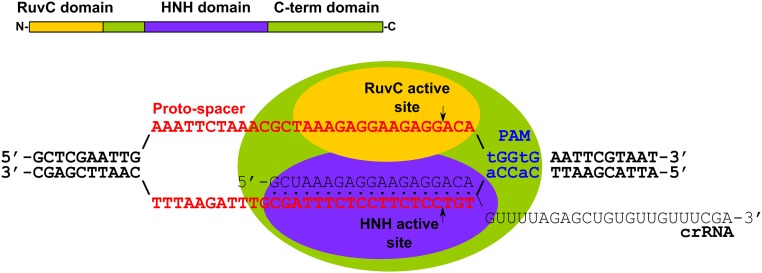
Comparación entre el nuevo coronavirus y brotes anteriores por otros CORONAVIRUS.
|
Coronavirus brotes |
Casos |
Fallecidos |
% |
|
MERS-CoV 2012 |
2.494 |
858 |
34,4% |
|
SARS-CoV 2003 |
8.098 |
774 |
9,6% |
|
Covid-19 2020 |
82.585 |
2.814 |
3,4% |
- El brote de SARS (SARS-CoV), Síndrome Respiratorio Agudo Severo, duró alrededor de ocho meses y murieron 774 personas de alrededor de 8.098 casos confirmados. 2003
- Síndrome Respiratorio de Oriente Medio (MERS-CoV,) 2012
- El nuevo coronavirus (Covid-19) ha superado la epidemia de SARS de 2003 en casos: confirmados y muertes
¿Cuáles son los síntomas de la infección POR CORONAVIRUS (Covid-19) ?
Los síntomas más comunes de COVID-19 son fiebre, cansancio y tos seca.
- Algunos pacientes pueden tener dolores y molestias, congestión nasal, secreción nasal, dolor de garganta o diarrea. Estos síntomas generalmente son leves y comienzan gradualmente.
- Algunas personas se infectan pero no desarrollan ningún síntoma y no se sienten mal.
- La mayoría de las personas (alrededor del 80%) se recuperan de la enfermedad sin necesidad de un tratamiento especial.
- Alrededor de 1 de cada 6 personas que contraen COVID-19 se enferma gravemente y desarrolla dificultad para respirar. Las personas mayores, y aquellas con problemas médicos subyacentes como presión arterial alta, problemas cardíacos o diabetes, tienen más probabilidades de desarrollar enfermedades graves.
- Las personas con fiebre, tos y dificultad para respirar deben buscar atención médica.
¿Cómo se propaga COVID-19?
Las personas pueden contraer COVID-19 de otras personas que tienen el virus. La enfermedad puede propagarse de persona a persona a través de pequeñas gotas de la nariz o la boca que se propagan cuando una persona con COVID-19 tose o exhala. Estas gotas caen sobre objetos y superficies alrededor de la persona. Luego, otras personas adquieren el COVID-19 al tocar estos objetos o superficies, luego se tocan los ojos, la nariz o la boca. Las personas también pueden contagiarse de COVID-19 si inhalan gotas de una persona con COVID-19 que tose o exhala gotas. Es por eso que es importante mantenerse a más de 1 metro de distancia de una persona enferma.
La OMS está evaluando la investigación en curso sobre las formas en que COVID-19 se propaga y continuará compartiendo resultados actualizados.
¿Se puede contraer CoVID-19 de una persona que no tiene síntomas?
La forma principal en que la enfermedad se propaga es a través de gotas respiratorias expulsadas por alguien que está tosiendo. El riesgo de contraer COVID-19 de alguien sin ningún síntoma es muy bajo. Sin embargo, muchas personas con COVID-19 experimentan solo síntomas leves. Esto es particularmente cierto en las primeras etapas de la enfermedad. Por lo tanto, es posible contraer COVID-19 de alguien que lo tiene, por ejemplo, con solo una tos leve y no se siente enfermo. La OMS está evaluando la investigación en curso sobre el período de transmisión de COVID-19 y continuará compartiendo resultados actualizados.
¿Puedo contraer COVID-19 de las heces de alguien con la enfermedad?
El riesgo de contraer COVID-19 de las heces de una persona infectada parece ser bajo. Si bien las investigaciones iniciales sugieren que el virus puede estar presente en las heces en algunos casos, la propagación a través de esta ruta no es una característica principal del brote. La OMS está evaluando la investigación en curso sobre las formas en que se disemina COVID-19 y continuará compartiendo nuevos hallazgos. Sin embargo, debido a que es un riesgo, es otra razón para lavarse las manos regularmente, después de usar el baño y antes de comer.
Medidas de protección para todos.
Limpie sus manos regularmente y a fondo con un desinfectante para manos a base de alcohol o láveselas con agua y jabón.
- ¿Por qué? Lavarse las manos con agua y jabón o usar un desinfectante para manos a base de alcohol mata los virus que pueden estar en sus manos.
Mantenga al menos 1 metro de distancia entre usted y cualquier persona que esté tosiendo o estornudando.
- ¿Por qué? Cuando alguien tose o estornuda, rocían pequeñas gotas de líquido de su nariz o boca que pueden contener virus. Si está demasiado cerca, puede respirar las gotas, incluido el virus COVID-19, si la persona que tose tiene la enfermedad.
Evite tocarse los ojos, la nariz y la boca.
- ¿Por qué? Las manos tocan muchas superficies y pueden detectar virus. Una vez contaminadas, las manos pueden transferir el virus a los ojos, la nariz o la boca. A partir de ahí, el virus puede ingresar a su cuerpo y enfermar.
Asegúrese de que usted y las personas que lo rodean sigan una buena higiene respiratoria. Esto significa cubrirse la boca y la nariz con el codo o pañuelo doblado cuando tose o estornuda. Luego deseche el tejido usado inmediatamente.
- ¿Por qué? Las gotas propagan el virus. Al seguir una buena higiene respiratoria, protege a las personas que lo rodean de virus como el resfriado, la gripe y COVID-19.
Quédese en casa si no se siente bien. Si tiene fiebre, tos y dificultad para respirar, busque atención médica y llame con anticipación. Siga las instrucciones de su autoridad de salud local.
- ¿Por qué? Las autoridades nacionales y locales tendrán la información más actualizada sobre la situación en su área. Llamar con anticipación permitirá que su proveedor de atención médica lo dirija rápidamente al centro de salud adecuado. Esto también lo protegerá y ayudará a prevenir la propagación de virus y otras infecciones.
Manténgase informado sobre los últimos desarrollos sobre COVID-19. Siga los consejos dados por su médico, su autoridad de salud pública local y nacional o su empleador sobre cómo protegerse y proteger a otros del COVID-19.
- ¿Por qué? Las autoridades nacionales y locales tendrán la información más actualizada sobre si COVID-19 se está extendiendo en su área. Están mejor ubicados para asesorar sobre lo que deberían hacer las personas en su área para protegerse.
Medidas de protección para personas que están o han visitado recientemente (en los últimos 14 días) áreas donde se está extendiendo COVID-19
Siga la guía descrita anteriormente. (Medidas de protección para todos)
Quédese en casa si comienza a sentirse mal, incluso con síntomas leves, como dolor de cabeza y secreción nasal leve, hasta que se recupere.
- ¿Por qué? Evitar el contacto con otras personas y las visitas a las instalaciones médicas permitirá que estas instalaciones funcionen de manera más efectiva y ayudará a protegerlo a usted y a otros de posibles COVID-19 y otros virus.
Si desarrolla fiebre, tos y dificultad para respirar, busque atención médica de inmediato ya que esto puede deberse a una infección respiratoria u otra afección grave. Llame con anticipación e informe a su proveedor de cualquier viaje reciente o contacto con viajeros.
- ¿Por qué? Llamar con anticipación permitirá que su médico lo dirija rápidamente al centro de salud adecuado. Esto también ayudará a prevenir la posible propagación de COVID-19 y otros virus.
¿Quién está en riesgo de desarrollar una enfermedad grave?
Si bien todavía estamos aprendiendo cómo COVID-2019 afecta a las personas, las personas mayores y las personas con afecciones médicas preexistentes (como enfermedad pulmonar, presión arterial alta, enfermedad cardíaca o diabetes) parecen desarrollar enfermedades graves con más frecuencia que otras.
¿COVID-19 es lo mismo que SARS?
No. El virus que causa COVID-19 y el que causa el Síndrome Respiratorio Agudo Severo (SRAS) están relacionados genéticamente entre sí, pero son diferentes. El SARS es más mortal pero mucho menos infeccioso que COVID-19. No ha habido brotes de SARS en ninguna parte del mundo desde 2003.
¿Debo usar una MASCARILLA para protegerme?
SU USO NO ESTÁ RECOMENDADO EN PERSONAS SANAS.
Las personas sin síntomas respiratorios, como la tos, no necesitan usar una máscara médica. La OMS recomienda el uso de máscaras para las personas que tienen síntomas de COVID-19 y para aquellos que cuidan a personas que tienen síntomas, como tos y fiebre. El uso de máscaras es crucial para los trabajadores de la salud y las personas que cuidan a alguien (en el hogar o en un centro de atención médica).
La OMS recomienda el uso racional de las máscaras médicas para evitar el desperdicio innecesario de recursos y el uso indebido de las máscaras (consulte el Consejo sobre el uso de máscaras). Use una máscara solo si tiene síntomas respiratorios (tos o estornudos), si sospecha una infección por COVID-19 con síntomas leves o si está cuidando a alguien con sospecha de infección por COVID-19. Una sospecha de infección con COVID-19 está relacionada con los viajes en áreas donde se han reportado casos, o el contacto cercano con alguien que ha viajado en estas áreas y ha enfermado.
Las formas más efectivas para protegerse y proteger a otros contra COVID-19 son limpiarse las manos con frecuencia, cubrirse la tos con la curva del codo o pañuelo y mantener una distancia de al menos 1 metro de las personas que están tosiendo o estornudando. Para obtener más información, consulte las medidas básicas de protección contra el nuevo coronavirus.
¿Cuánto dura el período de incubación de COVID-19?
El "período de incubación" significa el tiempo entre la invasión del virus y el comienzo de tener síntomas de la enfermedad. La mayoría de las estimaciones del período de incubación para COVID-19 varían de 1 a 14 días, más comúnmente alrededor de cinco días. Estas estimaciones se actualizarán a medida que haya más datos disponibles.
¿Pueden los humanos infectarse con el COVID-19 de una fuente animal?
Los coronavirus son una gran familia de virus que son comunes en los animales. En raras ocasiones, las personas se infectan con estos virus que luego pueden propagarse a otras personas.
Por ejemplo, el Coronavirus del SARS (SARS-CoV) fueidentificado en 2003. Se cree que el SARS-CoV es un virus animal, de un reservorio animal aún incierto, tal vez murciélagos, que se propagó a otros animales (gatos de civeta: pequeño mamífero, de hocico afilado y aspecto de gato, de la familia de la jineta) y a los primeros humanos infectados en el Provincia de Guangdong del sur de China en 2002. Y el MERS-CoV se transmite por camellos dromedarios .
Las posibles fuentes animales de COVID-19 aún no se han confirmado.
Para protegerse, como cuando visita mercados de animales vivos, evite el contacto directo con animales y superficies en contacto con animales. Garantizar buenas prácticas de seguridad alimentaria en todo momento. Manipule la carne cruda, la leche o los órganos animales con cuidado para evitar la contaminación de los alimentos crudos y evitar el consumo de productos animales crudos o poco cocidos.
¿Cuánto tiempo sobrevive el virus en las superficies?
No es seguro cuánto tiempo el virus que causa COVID-19 sobrevive en las superficies, pero parece comportarse como otros coronavirus. Los estudios sugieren que los coronavirus (incluida la información preliminar sobre el virus COVID-19) pueden persistir en las superficies durante algunas horas o hasta varios días. Esto puede variar bajo diferentes condiciones (por ejemplo, tipo de superficie, temperatura o humedad del ambiente).
Si cree que una superficie puede estar infectada, límpiela con un desinfectante simple para eliminar el virus y protegerse a sí mismo y a los demás. Lávese las manos con un desinfectante para manos a base de alcohol o lávelas con agua y jabón. Evite tocarse los ojos, la boca o la nariz.
¿Es seguro recibir un paquete de cualquier área donde se haya reportado COVID-19?
Sí. La probabilidad de que una persona infectada contamine productos comerciales es baja y el riesgo de contraer el virus que causa COVID-19 de un paquete que se ha movido, viajado y expuesto a diferentes condiciones y temperatura también es bajo.
¿Hay algo que no deba hacer?
Las siguientes medidas NO SON efectivas contra COVID-2019 :
- Tomar remedios herbales tradicionales
- Usar máscaras múltiples
- La automedicación con antibióticos
En cualquier caso, si tiene fiebre, tos y dificultad para respirar, busque atención médica temprana para reducir el riesgo de desarrollar una infección más grave y asegúrese de compartir su historial de viajes reciente con su médico.
FUENTE : Organización Mundial de la Salud 27 de febrero de 2020
RESUMIDO Y TRADUCIDO Y COMENTADO POR:
PROF. DR. FERNANDO GALAN