BREVE INTRODUCCIÓN
Hasta el 2022 se han atribuido, como causas de las Enfermedades Mitocondriales, a variantes patogénicas en más de 400 genes, tanto de origen mitocondrial como nuclear, de los aproximadamente 1500 genes que se estima que componen el proteoma mitocondrial del ADNn.
Causas de la Enfermedad mitocondrial:
- mutaciones primarias en el ADN mitocondrial: mutaciones puntuales y deleciones únicas. Herencia mitocondrial maternal
- mutaciones en el ADN nuclear: que dan como resultado defectos en las funciones de las mitocondrias, con patrón de herencia autosómico dominante, autosómico recesivo o ligado al cromosoma X, así como a deleciones múltiples o deplecion del ADN mitocondrial.
En el siguiente esquema se puede entender mejor lo referido anteriormente
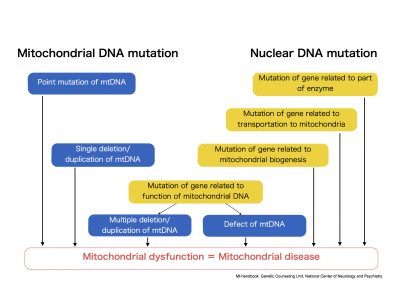
Mecanismos por lo que mutaciones en el ADN nuclear causan enfermedad mitocondrial:
- Múltiples deleciones de mtDNA
- Depleción de mtDNA
- Defectos de la subunidad de la cadena respiratoria (RC)
- Defecto de proteína auxiliar: defectos de proteína de ensamblaje,
- Defecto de síntesis de CoQ
- Defecto del metabolismo del hierro
- Defecto de motilidad mitocondrial
- Defecto de fusión mitocondrial
- Defecto de fisión mitocondrial
Secuencia de trabajo utilizada para identificar variantes asociadas con la enfermedad mitocondrial.
- En primer lugar, la información clínica es vital sospechar una enfermedad mitocondrial y para fundamentar las pruebas genéticas adecuadas.
- Recomendamos el uso de la secuenciación en trío mediante análisis simultáneo del probando (paciente) y de los padres, del exoma completo del ADN mitocondrial (WES) o del genoma completo del ADN nuclear (WGS).
ESTUDIOS GENÉTICOS EN PACIENTES CON SOSPECHA DE ENFERMEDAD MITOCONDRIAL
- Secuenciación del exoma del ADN mitocondrial o panel de Mutaciones patógenas del DNA mitocondrial confirmadas por Mitomap
Las que refiero aquí son las reportadas hasta el 11 de septiembre de 2021
|
Índice |
Tipo |
Gen |
Enfermedades asociadas |
Alelo mutado |
|
1 |
tRNA |
MT-TF |
MELAS / MM y EXIT |
|
|
2 |
tRNA |
MT-TF |
Epilepsia de herencia materna/enfermedad renal mitotubulointersticial (MITKD) |
|
|
3 |
tRNA |
MT-RNR1 |
DEAF |
|
|
4 |
tRNA |
MT-RNR1 |
Sordera; discapacidad intelectual del espectro autista; posiblemente antiaterosclerótico |
|
|
5 |
tRNA |
MT-TV |
AMDF |
|
|
6 |
tRNA |
MT-TV |
MNGIE-like disease / MELAS |
|
|
7 |
tRNA |
MT-TV |
Leigh Syndrome / HCM / MELAS |
|
|
8 |
tRNA |
MT-TL1 |
MELAS / Leigh Syndrome / DMDF / MIDD / SNHL / CPEO / MM / FSGS / ASD / Disfunción cardíaca + multiorgánica |
|
|
9 |
tRNA |
MT-TL1 |
MM / MELAS / SNHL / CPEO |
|
|
10 |
tRNA |
MT-TL1 |
MELAS; posible riesgo de aterosclerosis |
|
|
11 |
tRNA |
MT-TL1 |
MELAS / Miopatía |
|
|
12 |
tRNA |
MT-TL1 |
MMC / MELAS |
|
|
13 |
tRNA |
MT-TL1 |
MELAS / DM |
|
|
14 |
tRNA |
MT-TL1 |
PEM / distrofia retiniana en MELAS |
|
|
15 |
tRNA |
MT-TL1 |
Miopatía |
|
|
16 |
tRNA |
MT-TL1 |
MELAS / Myopathy / Sordera + Deterioro Cognitivo |
|
|
17 |
tRNA |
MT-TL1 |
MM |
|
|
18 |
tRNA |
MT-TL1 |
MMC |
|
|
19 |
Coding |
MT-ND1 |
LHON MELAS solapamiento o superposición |
|
|
20 |
Coding |
MT-ND1 |
LHON |
|
|
21 |
Coding |
MT-ND1 |
LHON |
|
|
22 |
Coding |
MT-ND1 |
MELAS / Leigh Syndrome / LDYT / BSN |
|
|
23 |
Coding |
MT-ND1 |
LHON |
|
|
24 |
Coding |
MT-ND1 |
LHON |
|
|
25 |
Coding |
MT-ND1 |
Progressive Encephalomyopathy / Leigh Syndrome / Optic Atrophy |
|
|
26 |
Coding |
MT-ND1 |
EXIT+mialgia/agrandamiento auricular izquierdo severo +insuficiencia cardiaca/aciduria 3-MGA |
|
|
27 |
Coding |
MT-ND1 |
LHON / Leigh-like phenotype |
|
|
28 |
tRNA |
MT-TI |
CPEO / MS. características clínicas tanto de la oftalmoplejía externa crónica progresiva como de la esclerosis múltiple |
|
|
29 |
tRNA |
MT-TI |
MICM |
|
|
30 |
tRNA |
MT-TI |
CPEO |
|
|
31 |
tRNA |
MT-TQ |
Encephalopathy / MELAS |
|
|
32 |
tRNA |
MT-TM |
Myopathy / MELAS / Leigh Syndrome |
|
|
33 |
tRNA |
MT-TW |
Mitochondrial myopathy |
|
|
34 |
tRNA |
MT-TW |
Leigh Syndrome |
|
|
35 |
tRNA |
MT-TA |
Myopathy |
|
|
36 |
tRNA |
MT-TN |
CPEO+ptosis+proximal myopathy |
|
|
37 |
tRNA |
MT-TN |
CPEO / MM |
|
|
38 |
tRNA |
MT-TN |
Fallo Multiorgánico / myopathy |
|
|
39 |
Coding |
MT-CO1 |
SNHL |
|
|
40 |
tRNA |
MT-TS1 precursor |
SNHL |
|
|
41 |
tRNA |
MT-TS1 |
PEM / AMDF / Similar a enfermedad de la neurona motora |
|
|
42 |
tRNA |
MT-TS1 |
MM / EXIT |
|
|
43 |
tRNA |
MT-TS1 |
SNHL |
|
|
44 |
tRNA |
MT-TS1 |
SNHL/Deafness |
|
|
45 |
tRNA |
MT-TK |
Severe adult-onset multisymptom myopathy / Myoclonic epilepsy |
|
|
46 |
tRNA |
MT-TK |
MNGIE / Progressive mito cytopathy |
|
|
47 |
tRNA |
MT-TK |
Myopathy / Exercise Intolerance / Eye disease+SNHL |
|
|
48 |
tRNA |
MT-TK |
MERRF; Other - LD / Depressive mood disorder / leukoencephalopathy / HiCM |
|
|
49 |
tRNA |
MT-TK |
MERRF |
|
|
50 |
tRNA |
MT-TK |
MICM+DEAF / MERRF / Autism / Leigh Syndrome / Ataxia+Lipomas |
|
|
51 |
Coding |
MT-ATP8/6 |
Infantile cardiomyopathy |
|
|
52 |
Coding |
MT-ATP6 |
BSN / Leigh syndrome |
|
|
53 |
Coding |
MT-ATP6 |
Mitochondrial myopathy, lactic acidosis and sideroblastic anemia (MLASA) / IgG nephropathy |
|
|
54 |
Coding |
MT-ATP6 |
NARP / Leigh Disease / MILS / other |
|
|
55 |
Coding |
MT-ATP6 |
NARP / Leigh Disease / MILS / other |
|
|
56 |
Coding |
MT-ATP6 |
Ataxia syndromes |
|
|
57 |
Coding |
MT-ATP6 |
MIDD, renal insufficiency |
|
|
58 |
Coding |
MT-ATP6 |
FBSN / Leigh Disease |
|
|
59 |
Coding |
MT-ATP6 |
Leigh Disease / Spastic Paraplegia |
|
|
60 |
Coding |
MT-ATP6 |
Leigh Disease / Ataxia syndromes / NARP-like disease |
|
|
61 |
Coding |
MT-ATP6 |
Encephalopathy / Seizures / Lacticacidemia |
|
|
62 |
tRNA |
MT-TG |
PEM |
|
|
63 |
Coding |
MT-ND3 |
Leigh Disease / MELAS |
|
|
64 |
Coding |
MT-ND3 |
Leigh Disease / Leigh-like Disease / ESOC |
|
|
65 |
Coding |
MT-ND3 |
Leigh Disease / Dystonia / Stroke / LDYT |
|
|
66 |
Coding |
MT-ND4L |
LHON |
|
|
67 |
Coding |
MT-ND4 |
Leigh Disease |
|
|
68 |
Coding |
MT-ND4 |
LHON / Progressive Dystonia |
|
|
69 |
tRNA |
MT-TH |
MERRF-MELAS / Encephalopathy |
|
|
70 |
tRNA |
MT-TH |
Maternally inherited non-syndromic deafness |
|
|
71 |
tRNA |
MT-TS2 |
DMDF / RP+SNHL |
|
|
72 |
tRNA |
MT-TL2 |
CPEO |
|
|
73 |
tRNA |
MT-TL2 |
CPEO / EXIT+Ophthalmoplegia |
|
|
74 |
tRNA |
MT-TL2 |
CPEO / KSS / possible carotid atherosclerosis risk, trend toward myocardial infarction risk |
|
|
75 |
tRNA |
MT-TL2 |
CPEO |
|
|
76 |
Coding |
MT-ND5 |
Leigh Disease |
|
|
77 |
Coding |
MT-ND5 |
Optic neuropathy/ retinopathy/ LD |
|
|
78 |
Coding |
MT-ND5 |
LHON |
|
|
79 |
Coding |
MT-ND5 |
Ataxia+PEO / MELAS, LD, LHON, myoclonus, fatigue |
|
|
80 |
Coding |
MT-ND5 |
LHON |
|
|
81 |
Coding |
MT-ND5 |
Leigh Disease / MELAS / LHON-MELAS Overlap Syndrome / negative association w Carotid Atherosclerosis |
|
|
82 |
Coding |
MT-ND5 |
Leigh Disease / MELAS / Ca2+ downregulation |
|
|
83 |
Coding |
MT-ND6 |
LDYT / Leigh Disease / dystonia / carotid atherosclerosis risk |
|
|
84 |
Coding |
MT-ND6 |
LHON |
|
|
85 |
Coding |
MT-ND6 |
LHON |
|
|
86 |
Coding |
MT-ND6 |
LHON |
|
|
87 |
Coding |
MT-ND6 |
Dystonia / Leigh Disease / ataxia / ptosis / epilepsy |
|
|
88 |
Coding |
MT-ND6 |
LHON |
|
|
89 |
Coding |
MT-ND6 |
LHON |
|
|
90 |
tRNA |
MT-TE |
Reversible COX deficiency myopathy |
|
|
91 |
tRNA |
MT-TE |
MM+DMDF / Encephalomyopathy / Dementia+diabetes+ophthalmoplegia |
|
|
92 |
tRNA |
MT-TE |
Encephalomyopathy + Retinopathy |
|
|
93 |
Coding |
MT-CYB |
EXIT / Septo-Optic Dysplasia |
|
|
94 |
Coding |
MT-CYB |
Multisystem Disorder, EXIT |
|
|
95 |
tRNA |
MT-TP |
MM / PEO |
Abreviaturas: AMDF, ataxia, mioclono y sordera; ATP6, ATP sintasa F0 subunidad 6; BSN = necrosis estriatal bilatera; CPEO, oftalmoplejía externa progresiva crónica; CYB, citocromo b; DEAF, sordera heredada de la madre o sordera inducida por aminoglucósidos; DM, diabetes mellitus; DMDF, diabetes mellitus+sordera; EXIT, intolerancia al ejericio; FBSN, necrosis estriatal bilateral familiar; KSS, síndrome de Kearns-Sayre; LDYT: neuropatía óptica hereditaria de Leber y DYsTonia; MELAS, encefalopatía mitocondrial, miopatía, acidosis láctica y episodios tipo ictus; MERRF, epilepsia mioclónica y fibras rojas rasgadas; MICM, miocardiopatía de herencia materna; MILS, síndrome de Leigh de herencia materna; MM, miopatía mitocondrial; MMC, miopatía materna y miocardiopatía; PEM, encefalopatía progresiva; RP, retinitis pigmentosa
- Secuenciación del genoma (whole genome sequencing o WGS) de un panel de genes DEL ADN NUCLEAR asociados a enfermedades mitocondriales. Aquí hago referencia a un panel de 259 genes.
Si no se identifican variantes patogénicas del ADN nuclear en el panel se recomienda secuenciación del genoma completo del ADN nuclear (WGS).
Genes nucleares directamente relacionados con la biogénesis de la fosforilación oxidativa que se han relacionado con enfermedades mitocondriales.
|
Fosforilación oxidativa (OXPHOS), Cadena de transporte de electrones (ETC) subunidades |
|
|
Complejo I |
|
|
Subunidades con codificación nuclear |
NDUFA1, NDUFA2, NDUFA9, NDUFA10, NDUFA11, NDUFA12, NDUFA13, NDUFB3, NDUFB9, NDUFB10, NDUFB11, NDUFS1, NDUFS2, NDUFS3, NDUFS4, NDUFS6, NDUFS7, NDUFS8, NDUFV1, NDUFV2 20 |
|
Complejo II |
|
|
Subunidades con codificación nuclear |
SDHA, SDHB, SDHD 3 |
|
Complejo III |
|
|
Subunidades con codificación nuclear |
CYC1, UBCRB, UQCRC2 3 |
|
Complejo IV |
|
|
Subunidades con codificación nuclear |
COX41, COX412, NDUFA4 3 |
|
Complejo V |
|
|
Subunidades con codificación nuclear |
ATP5A1, ATP5E 2 |
|
Factores de ensamblaje de OXPHOS |
|
|
Complejo I |
|
|
Genes con codificación nuclear |
ACAD9, FOXRED1, NDUFAF1, NDUFAF2, NDUFAF3, NDUFAF4, NDUFAF5, NDUFAF6, NUBPL, TIMMDC1, TIMEM126B 11 |
|
Complejo II |
|
|
Genes con codificación nuclear |
SCHAF1 1 |
|
Complejo III |
|
|
Genes con codificación nuclear : |
BCSIL, LYRM7, TTC19, UQCC2 4 |
|
Complejo IV |
|
|
Genes con codificación nuclear |
COA3, COA5, COA6, COA7, COX10, COX14, SCO1, SCO2, COX15, COX20, PET100, APOFT1, SURF1, PET 117 14 |
|
Complejo V |
|
|
Genes con codificación nuclear : |
ATPAF2, TMEM70, USMG5 3 |
|
Replicación/homeostasis, transcripción, traducción |
|
|
replicación/homeostasis del mtDNA |
|
|
Genes con codificación nuclear |
POLG, POLG2, DNA2, MGME1, RNASEH1, TFAM, TWNK 7 |
|
Conjunto de Nucleotidos |
|
|
Genes con codificación nuclear |
ABAT, DGUOK, MPV17, RRM2B, SAMHD1, SUCLA2, SUCLG1, TK2, TYMP 9 |
|
Portadores de electrones |
|
|
Genes con codificación nuclear |
COQ2, COQ4, COQ5, COQ6, COQ7, COQ8A, COQ9, PDSS1, PDSS2, CYCS, HCCS 11 |
|
Biogénesis de mt-ARNt |
|
|
Genes con codificación nuclear |
GTPP3, MTFMT, NSUN3, PUS1, ORSL1, TRIT1, TRMT5, TRMU, TRNT1, MTO1 10 |
|
Aminoacilación de mt-ARNt |
|
|
Genes con codificación nuclear |
AARS2, CARS2, DARS2, FARS2, GARS, HARS2, IARS2, KARS2, LARS2, MARS2, NARS2, PARS2, RARS2, SARS2, TARS2, VARS2, WARS2, YARS2 18 |
|
Expresión/procesamiento de mtRNA |
|
|
Genes con codificación nuclear |
ELAC2, FASTKD2, HSD17810, LRPPRC, MRM2, MTPAP, PNPT1, TRMG10C 8 |
|
Biosíntesis de ribosomas mitocondriales |
|
|
Genes con codificación nuclear |
MRPL12, MRPL44, MRP57, MRPS16, MRPS22, MRPS23, MRPS34, ERAL1, MMPL3 9 |
|
Traducción |
|
|
Genes con codificación nuclear |
C12orf65, GRM1, GFM2, RMD1, TSFM, TUFM, TAC 7 |
Genes con codificación nuclear involucrados en la fisiología mitocondrial que no están directamente relacionados con la fosforilación oxidativa que induce la enfermedad mitocondrial
|
Biosíntesis del grupo Fe-SFe-S |
ABCB7, BOLA3, FDX1L, FDXR, FXN, GLRX5, IBA57, ISCA2, ISCU, LYRM4, NFS1, NFU1, IREB2, C19orf12 14 |
|
Cofactores de enzimas |
COASY, FLAD1, LIAS, LIPT1, PANK2 5 |
|
Control de calidad de proteínas: |
AFG3L2, CLPP, LONP1, SPG7, YME1L1, PARL, PMPCB, IMMP2L, HTRA2, XPNPEP3 10 |
|
Modificación de lípidos: |
AAD3A, CHKB, PLA2G6, SERAC1, TAZ 5 |
|
Importación/procesamiento de proteínas |
AKG, AIFM1, DNAJC19, GFER, MIPEP, PMPCA, TIMM8A, TIMM50, AMT, GLDC 10 |
|
Morfología mitocondrial: |
C19orf70, DNM1L, GDAP1, MFF, MFN2, MSTO1, OPA1, SACS, SLC25A46, STAT2, TRANK1, VPS13D, VPS13A 13 |
|
Metabolismo Matriz mitocondrial |
D2HGDH, ECHS1, ETHE1, HIBCH, L2HGDH, NAKE, TXN2 7 |
|
Transporte metabólico |
SLC19A1, SLC25A1, SLC25A3, SLC25A4, SLC25A10, SLC25A12, SLC25A13, SLC25A15, SLC25A16, SLC25A19, SLC25A20, SLC25A21, SLC25A22, SLC25A24, SLC25A26, SLC25A32, SLC25A38, SLC25A42, SLC25A46, SLC2A13, MICU1, MICU2, MPC1 |
|
Ciclo y metabolismo del ácido tricarboxílico (TCA), o ciclo de Krebs |
ACO2, ALDH18A1, DLAT, DLD, FH, HAAO, IDH3A, IDH3B, KYNU, MDH2, MECR, NADK2, PDHA1, PDHB, PDHX, PDK3, PDP1, PPA2 |
|
Apoptosis/autofagia |
HTRA2, VPS13C |
|
Función poco clara: |
APOPT1, C19orf12, C1QBP, FBXL4, OPA3, RTN4IP1, SFXN4, TMEM65, CYP2U1 |
- Estudios genéticos de deleciones únicas, múltiples o depleción del ADNmt:
- PCR de amplio rango para la detección de deleción única o múltiples y depleción del ADN mitocondrial. En muestra de biopsia muscular
- Amplificación de sonda dependiente de ligación múltiple (MLPA) en leucocitos de sangre periférica con 23 paneles
Pruebas que identifican deleciones/duplicaciones que no se detectan fácilmente mediante análisis de secuencias. Los métodos utilizados pueden incluir PCR cuantitativa, PCR de largo alcance, amplificación de sonda dependiente de ligadura multiplex (MLPA) y transferencia de Southern, que es la técnica utilizada tradicionalmente.
Las Deleciones varían en tamaño de 1,1 a 10 kb. Las duplicaciones rara vez ocurren con deleciones, pero pueden ocurrir solas. Hasta la fecha, se han descrito más de 800 deleciones de ADNmt humano en la literatura científica y se han recopilado y organizado en la base de datos en línea MitoBreak.
Pueden heredarse de la madre o pueden aparecer de novo. La presencia de deleciones múltiples suele apuntar a defectos en los genes nucleares, que tienen una función importante en la replicación y el mantenimiento del mtDNA (POLG, POLG2, SLC25A4, PEO1 y OPA1). La depleción de mtDNA se puede detectar mediante PCR en tiempo real, que también es indicativo de mutaciones genéticas nucleares.
COMENTARIOS
La mayoría de los médicos de cabecera atenderán en algún momento a un paciente que “no es posible que haya tantas cosas mal en ella o en él”; “debe estar todo en sucabeza” o incluso a veces se les dice “que no tienen enfermedad y son hipocondríaca/os”. Vuelven repetidamente con síntomas que simplemente no parecen coincidir con su apariencia externa o si son reales, y los análisis y pruebas son negativas. Los pacientes suelen terminar con múltiples diagnósticos patológicos erróneos, que se suma a una mayor confusión. El médico a menudo se siente impotente para comprenderlos claramente.
Las mutaciones del ADN mitocondrial están presentes en una de cada 200 personas (llamadas portadoras) y al menos una de cada 4.700 padece la enfermedad. Eso significa que en una práctica general de más de 2,000 pacientes de cupo, aproximadamente 10 pacientes pueden ser portadores de una mutación que tiene el potencial de causar una enfermedad mitocondrial.
¿CUÁNDO SOSPECHAR UNA ENFERMEDAD MITOCONDRIAL EN PERSONAS CON SÍNTOMAS INICIALES INESPÉCIFICOS?
- Sospechar una enfermedad mitocondrial EN ADULTOS con estos síntomas iniciales inespecíficos como: debilidad muscular, cansancio crónico e intolerancia al ejercicio. Las 3 principales especialidades médicas visitadas por los pacientes adultos fueron medicina interna (15 %), medicina general (10 %) y medicina familiar (9 %).
- Sospechar una enfermedad mitocondrial en EDAD PEDIÁTRICA con estos síntomas iniciales inespecíficos como retraso en el desarrollo, debilidad muscular y epilepsia. Las 3 principales especialidades médicas visitadas por pacientes pediátricos fueron pediatría (24 %), práctica general (9 %) y neurología (6 %).
El promedio de médicos consultados hasta llegar al diagnóstico suele ser de unos 8 aproximadamente.
¿Cuándo es apropiado considerar a las mitocondrias como culpables y cómo podemos siquiera comenzar a investigarlo? Estos pacientes generalmente acuden a medicina general y/o medicina familiar– si no se les ha colocado ya la etiqueta por algún médico de la Seguridad Social o privado de padecer DEPRESIÓN, FIBROMIALGIA, SINDROME DE FATIGA CRÓNICA o SÍNDROME DEL DOLOR CENTRAL – son remitidos al Servicio de Neurología y dentro de este, a la sección de Enfermedades Neuromusculares. Donde algunos de los distintos especialistas, no les reciben con especial atención porque se refugian en que es una enfermedad que no tiene cura y muchos de ellos como mucho les hacen un hemograma, un perfil bioquímico y análisis basal de lactato, un EMG y después quizá una biopsia muscular.
El diagnóstico de la enfermedad mitocondrial se ha basado tradicionalmente en una biopsia de tejido invasiva para el análisis bioquímico e histoquímico, que puede ser normal incluso en pacientes con un diagnóstico genético definido.
Razones que conducen a un diagnóstico genético no concluyente.
Se podría plantear la hipótesis de que al menos una fracción de los casos no diagnosticados podría deberse a la interrupción de los elementos funcionales en las regiones no codificantes: regiones reguladoras, como promotores, potenciadores, elementos estructurales por un lado y ncRNA, así como micropéptidos ocultos. dentro de ncRNAs, por el otro.
Sin embargo, todavía NO es posible realizar un diagnóstico genético en más del 40% de los casos, incluso en cohortes muy seleccionadas, de ahí la necesidad de nuevos enfoques.
- Otras investigaciones que probablemente aumenten el rendimiento diagnóstico más allá de WGS incluyen estudios de laboratorio de la función mitocondrial y otros enfoques "ómicos", incluida la transcriptómica, que proporcionó un diagnóstico adicional en el 10 % de los pacientes con sospecha de enfermedad mitocondrial.
El rendimiento diagnóstico es superior con la técnica secuenciación del genoma completo (WGS) que con la secuenciación del exoma completo (WES), y es el resultado de su capacidad no solo para identificar las variantes de diagnóstico detectables por WES sino también para detectar variantes de diagnóstico más allá del alcance de WES. La ventaja de usar WGS en lugar de WES fue que es más fácil detectar variantes del número de copias (CNV), expansiones repetidas y heteroplasmias de bajo nivel. Porque WES no los detecta.
- WGS (secuenciación del genoma completo) analiza todas las regiones codificantes y no codificantes del genoma.
- WES (secuenciación de todo el exoma) se dirige solo a los exones codificantes más los límites inmediatos entre intrón y exón.
- La captura de genes: facilita la secuenciación de una lista de genes de enfermedades candidatos
Con el tiempo, mejorará la capacidad de interpretar intrónicos profundos y otras variantes de WGS no codificantes, aumentando así la ventaja diagnóstica de WGS sobre WES. Nuestra predicción, por lo tanto, es que las desventajas percibidas de WGS en relación con WES, incluido el aumento de costos y mayores requisitos para el análisis y almacenamiento de datos, serán finalmente superadas por su superioridad diagnóstica.
Los estudios genéticos clínicos de rutina en el diagnóstico de enfermedad mitocondrial en grandes cohortes tienen un rendimiento diagnóstico más bajo (34,5-39 %), lo que se cree que refleja más la práctica clínica diaria.
El enfoque actual para el diagnóstico de enfermedad mitocondrial, incluso con poca sospecha, es estudiar mutaciones del mtDNA mediante WES o un panel de genes mitocondriales específicos, actualmente 96 mutaciones. Y si fuera negativo, estudiar mutaciones el ADN nuclear mediante WGS.
Por lo tanto, muchos médicos que practican la medicina mitocondrial se preguntan ¿sí la biopsia muscular sigue siendo el estándar de oro para el diagnóstico de la enfermedad mitocondrial? NO LO ES. Los estudios de biopsia muscular solo deben utilizarse en situaciones clínicamente urgentes o para confirmar un resultado genético no concluyente.
Además, los defectos en el mantenimiento mitocondrial, fusión / fisión, traducción, transcripción o anomalías en la integridad o el transporte de la membrana mitocondrial nos han enseñado que algunos de estos trastornos NO siempre se presentan con deficiencias significativas de OXPHOS y pueden presentarse con resultados bioquímicos e histológicos musculares normales.
CONCLUSIONES
- El enfoque inicial actual para el diagnóstico de enfermedad mitocondrial, INCLUSO CON BAJA SOSPECHA, ES GENÉTICO. SOSPECHAR una enfermedad mitocondrial en EDAD PEDIÁTRICA con estos síntomas iniciales inespecíficos como: retraso en el desarrollo, debilidad muscular y epilepsia. Y SOSPECHAR una enfermedad mitocondrial EN ADULTOS en adultos con estos síntomas iniciales inespecíficos como: debilidad muscular, cansancio crónico e intolerancia al ejercicio.
- ODISEA DIAGNÓSTICA: estos pacientes generalmente acuden a medicina general y medicina familiar– si no se les ha colocado ya la etiqueta por algún médico de la Seguridad Social o privado de padecer DEPRESIÓN, FIBROMIALGIA, SINDROME DE FATIGA CRÓNICA o SÍNDROME DEL DOLOR CENTRAL – son remitidos al Servicio de Neurología y dentro de este, a la sección de Enfermedades Neuromusculares. Donde algunos de los distintos especialistas, no les reciben con especial atención porque se refugian en que es una enfermedad que no tiene cura y como mucho les hacen un hemograma, un perfil bioquímico y análisis basal de lactato, un EMG y después quizá una biopsia muscular.
- El diagnóstico de la enfermedad mitocondrial se ha basado tradicionalmente en una biopsia de tejido invasiva para el análisis bioquímico e histoquímico, QUE PUEDE SER NORMAL INCLUSO EN PACIENTES CON UN DIAGNOSTICO GENÉTICO DEFINIDO.
- Muchos médicos que practican la medicina mitocondrial se preguntan - SÍ LA BIOPSIA MUSCULAR SIGUE SIENDO EL ESTÁNDAR DE ORO PARA EL DIAGNÓSTICO DE LA ENFERMEDAD MITOCONDRIAL. PUES NO LO ES. Los estudios de biopsia muscular solo deben utilizarse en situaciones clínicamente urgentes o para confirmar un resultado genético no concluyente.
REFERENCIA
- Saneto RP. Mitochondrial diseases: expanding the diagnosis in the era of genetic testing. J Transl Genet Genom 2020;4:[Online First]. http://dx.doi.org/10.20517/jtgg.2020.40
- Gusic M, Prokisch H. Genetic basis of mitochondrial diseases. FEBS Lett. 2021 Apr;595(8):1132-1158. doi: 10.1002/1873-3468.14068. Epub 2021 Mar 21. PMID: 33655490.
- Alston CL, Stenton SL, Hudson G, Prokisch H, Taylor RW. The genetics of mitochondrial disease: dissecting mitochondrial pathology using multi-omic pipelines. J Pathol. 2021 Jul;254(4):430-442. doi: 10.1002/path.5641. Epub 2021 Mar 26. PMID: 33586140; PMCID: PMC8600955.
PROF. DR. FERNANDO GALAN